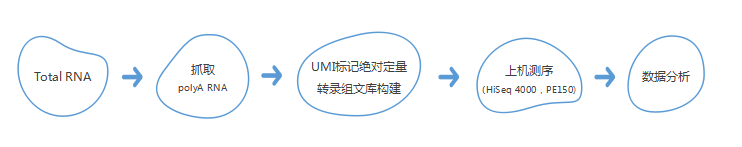
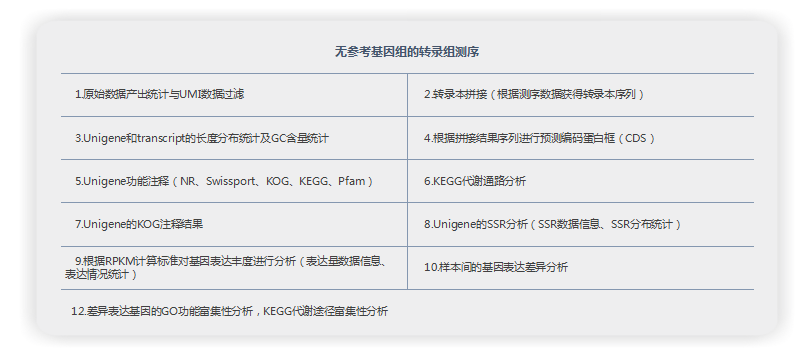
常规转录组测序由于文库PCR扩增偏好性,所有序列并不会被同比例放大,因而造成测序定量结果与样本中转录本的原始丰度不一致,导致差异基因筛选不准确(图1)。这也是造成qPCR验证测序结果不一致的主要原因。联川生物推出的绝对定量转录组测序,采用主流UMI标记技术[1,2,3],通过UMI标记每一条序列,可以消除PCR扩增偏好对定量的干扰,真实反映样本中转录本的表达丰度(图2)。在文库PCR扩增和测序过程中难免会引入错误的碱基,具有相同UMI标记的序列可以基于多序列比对来纠正PCR扩增和测序过程中引入的错误,确保获得转录本的真实序列(见图3)。
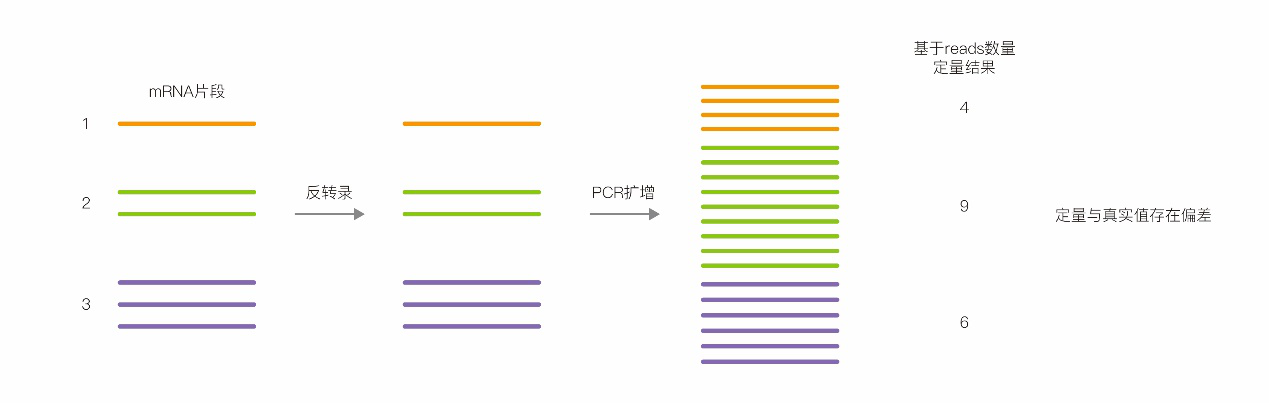
图1 PCR扩增产生的duplication显著干扰准确定量
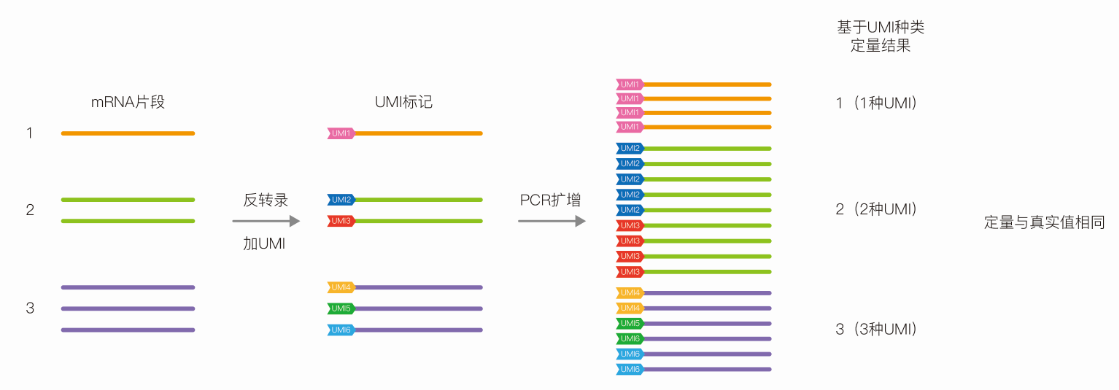
图2 UMI标记技术消除duplication干扰
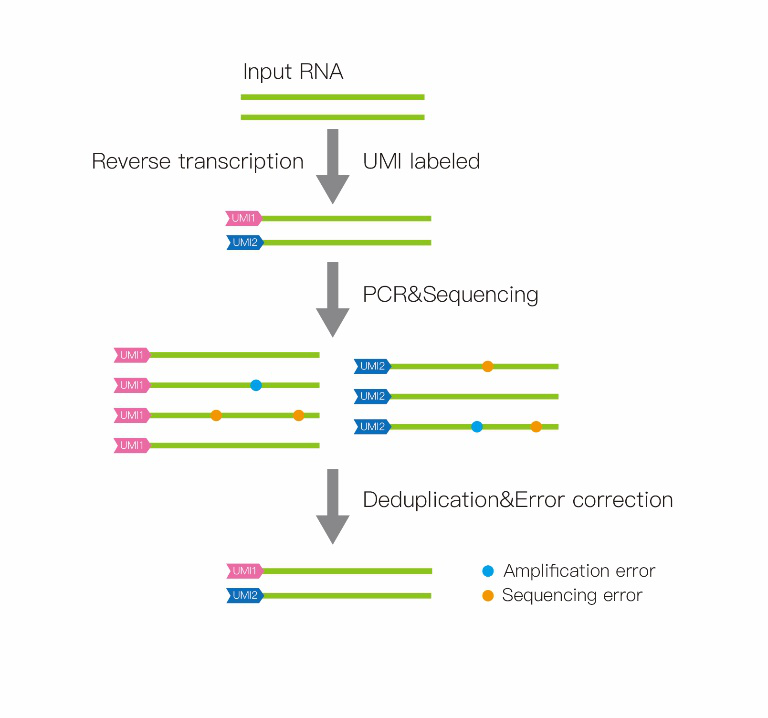
图3 UMI标记技术纠正序列错误
真实定量:采用UMI标记技术,每一条序列都被唯一标识,保留序列真重复,去除PCR扩增假重复,真实反映样本中转录本的表达丰度。真实序列:具有相同UMI标记的序列可以基于多序列比对来纠正PCR扩增和测序过程中引入的错误, 确保获得转录本的真实序列。

细胞,组织,全血,血清,血浆,总RNA等
建议总RNA起始量:2 μg,最低1 μg,浓度≥50 ng/μL
参考文献
1.Shiroguchi K, et al. Digital RNA sequencing minimizes sequence-dependent bias and amplification noise with optimized single-molecule barcodes. Proc Natl Acad Sci U S A. 2012 Jan 24;109(4):1347-52.2.Kivioja T, et al. Counting absolute numbers of molecules using unique molecular identifiers. Nat Methods. 2011, 9(1):72-43.Saiful Islam, et al. Quantitative single-cell RNA-seq with unique molecular identifiers. Nature Methods 2014, 11:163-166.
A1:主要有这6个原因:1)文库PCR扩增偏好,2)你验证的分子不对,3)用不同批次的样品做验证,4)用不同的样本类型做验证,5)拿低表达或差异不显著的基因做验证,6)样本比对关系搞反。查看详情
A2:使用绝对定量转录组测序,助您筛选出真实的差异表达基因,让你的时间不再花在假阳性结果的验证上,更快地开展下游功能验证实验,快人一步地发表研究成果。
这项工作讨论了如何最小化PCR扩增偏好性(就是Duplication)对测序数据,特别是低拷贝序列,定量分析的干扰。研究团队开发出了一种称为Digital RNA sequencing的方法:序列在反转录后,加入大量的标签(barcode),几乎每个cDNA都被唯一的barcode标记,然后进行PCR扩增获得转录组测序文库(见下图)。由于序列是被barcode唯一标记的,计算序列拷贝数时,不再直接统计同一种reads的数量,而是统计每种reads有多少个unique的barcode。而具有相同barcode的同一种reads,无论有多少拷贝数,都只计作一个拷贝,即来自PCR扩增的假重复(Duplication)被有效去除。
可以简单地认为,在样本中cDNA1有3个拷贝,cDNA2有2个拷贝,比例为3:2,然后用大量不同的barcode标记这5条序列,每条序列都被unique的barcode标记,最后进行PCR扩增完成文库制备。由于Duplication的存在,在没有barcode标记的情况下,cDNA1经扩增变成了9个拷贝,cDNA2变成了12个拷贝,比例变成了3:4,而两者原始的比例是3:2;而标记了barcode的cDNA1和cDNA2,合并具有相同barcode的同种序列后,cDNA1和cDNA2仍保持原始的拷贝数和比例,此种方案下测序定量结果准确反映了样本中序列的真实丰度和比例。

图 标签标记序列法去除Duplication的原理
Shiroguchi K, et al. Digital RNA sequencing minimizes sequence-dependent bias and amplification noise with optimized single-molecule barcodes. Proc Natl Acad Sci U S A. 2012 Jan 24;109(4):1347-52.
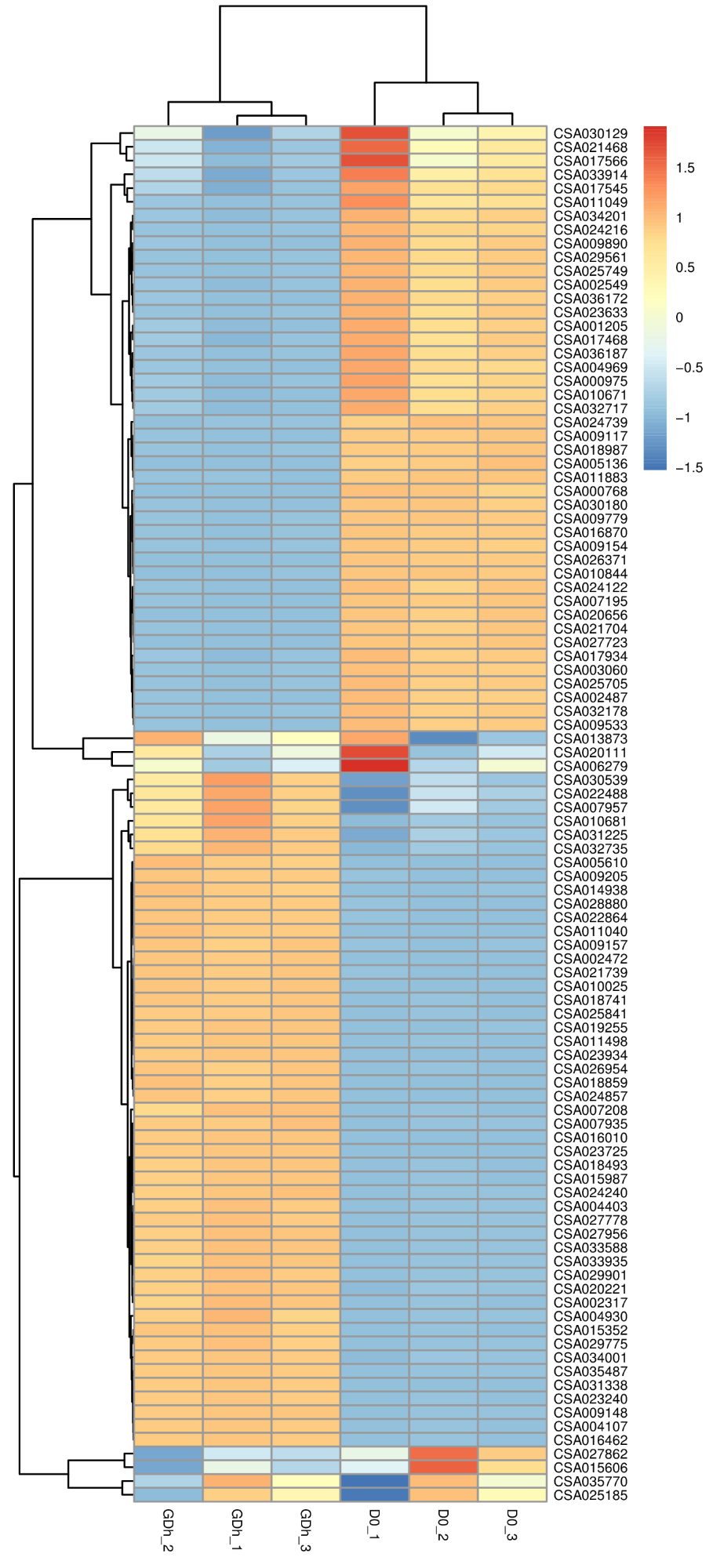
差异基因聚类分析热图
用于判断基因在不同实验条件下调控模式的聚类模式根据样品基因表达谱的相近程度,将基因进行聚类分析,直观地展示基因在不同样品(或是不同处理)中的表达情况,由此获取生物学相关信息。
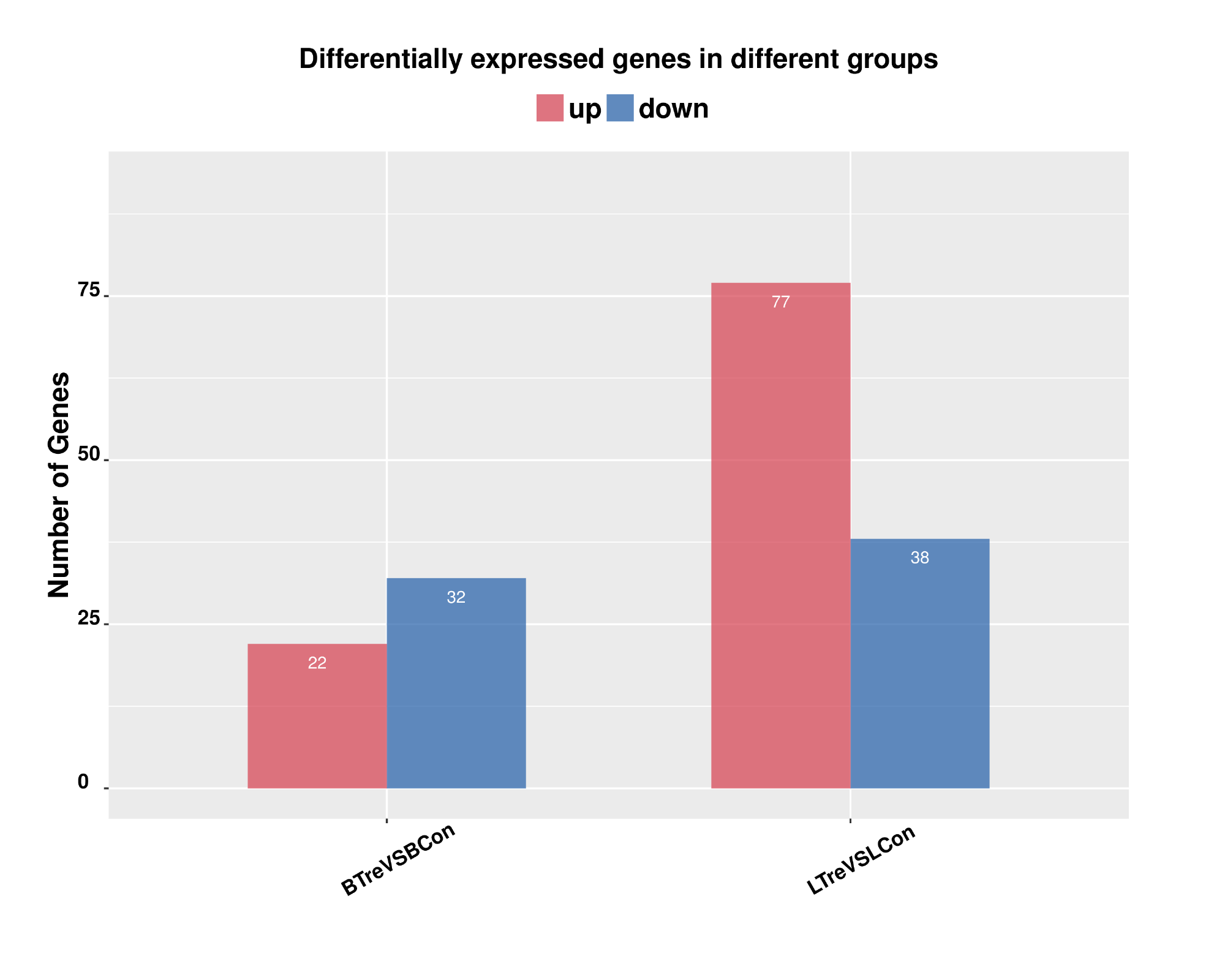
差异显著差异表达基因上下调频数统计柱状图用于统计差异基因数目
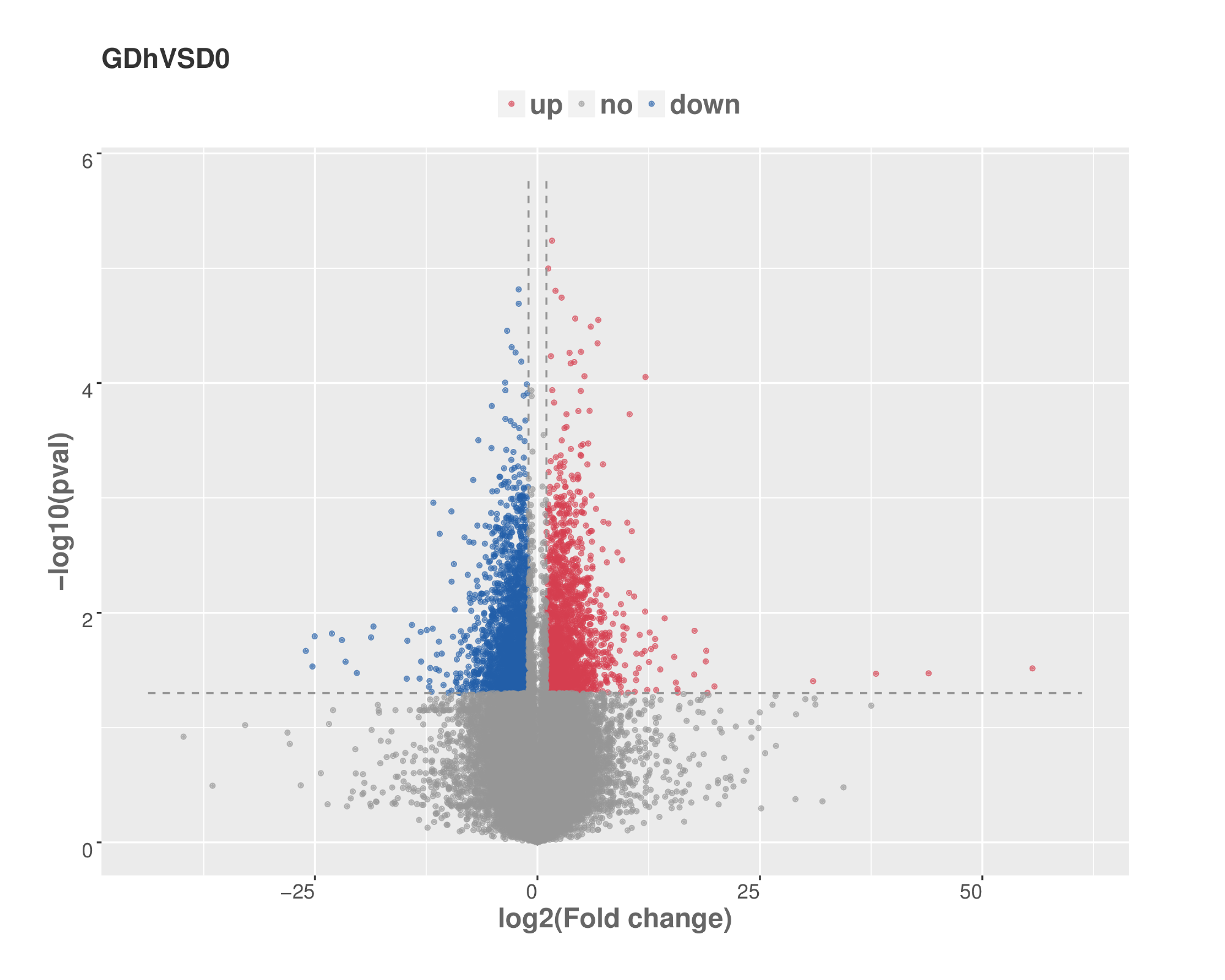
差异表达基因分析火山图
用于了解差异表达基因的整体分布情况。以log2(foldchange)为横坐标,-log10(pvalue)为纵坐标,对差异表达分析中所有的基因绘制火山图。其中横坐标代表基因在不同样本中差异表达倍数变化;纵坐标代表基因表达量变化差异的统计学显著性。
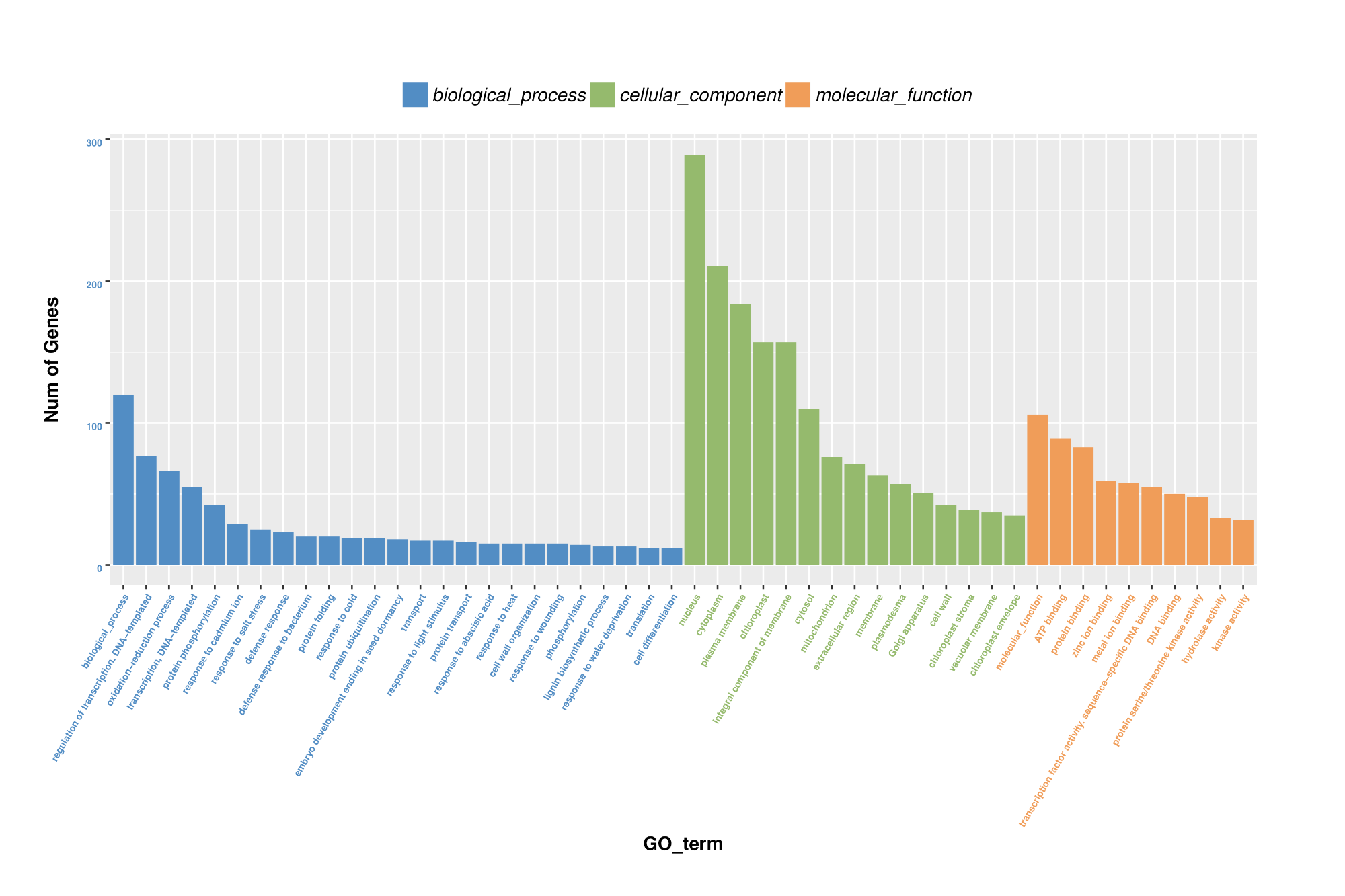
差异基因GO富集柱状图
用于反映在生物过程(biological process)、细胞组分(cellular component)和分子功能(molecular function)富集的GO term上差异基因的个数分布情况。
GO功能富集散点图
用于展示GO term的富集情况。Rich factor表示位于该GO的差异基因个数/位于该GO的总基因数,Rich factor越大,GO富集程度越高。

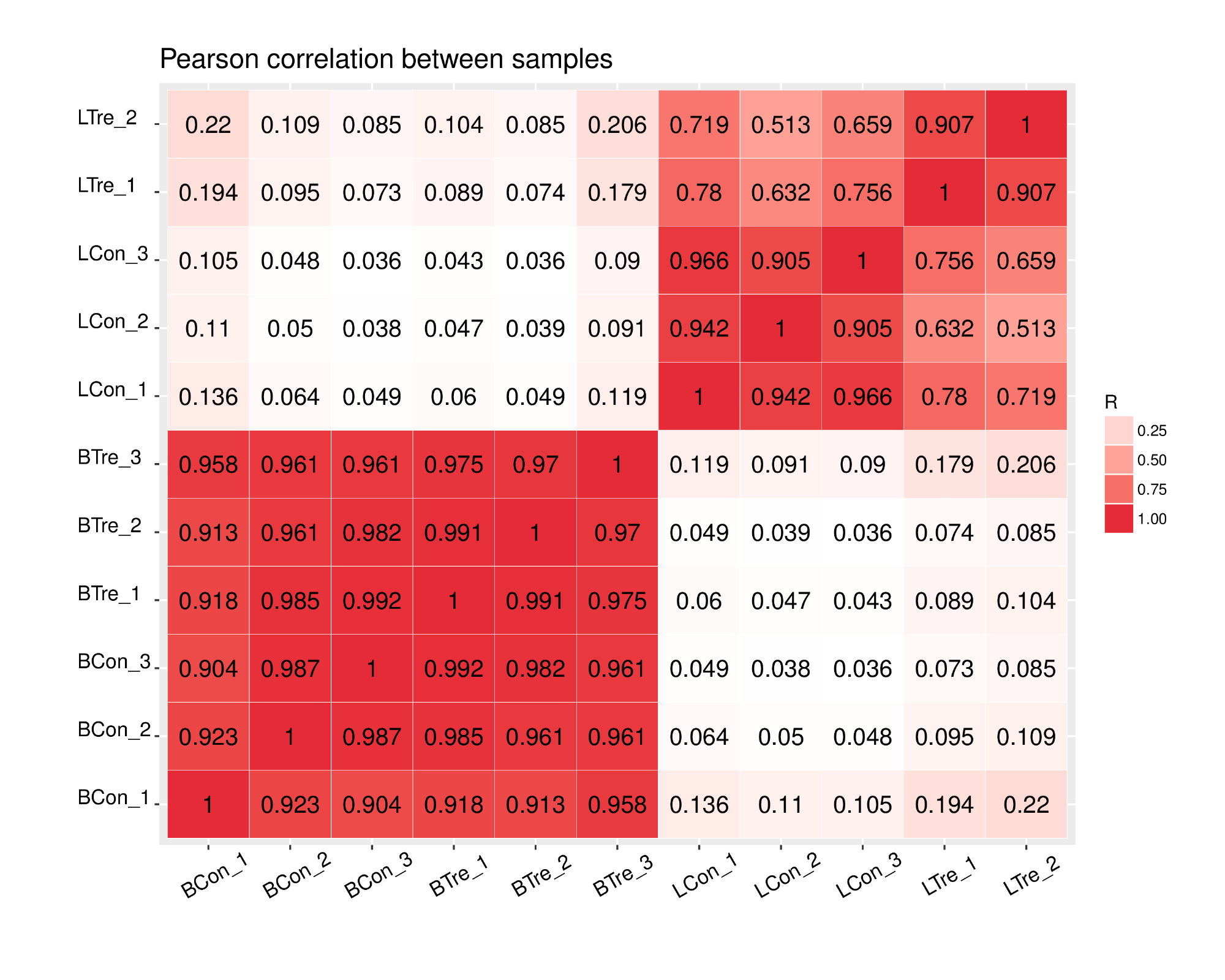
皮尔森相关系数图
用于反映生物学重复、样本相关性,确保后续的差异基因分析得到更可靠的结果。
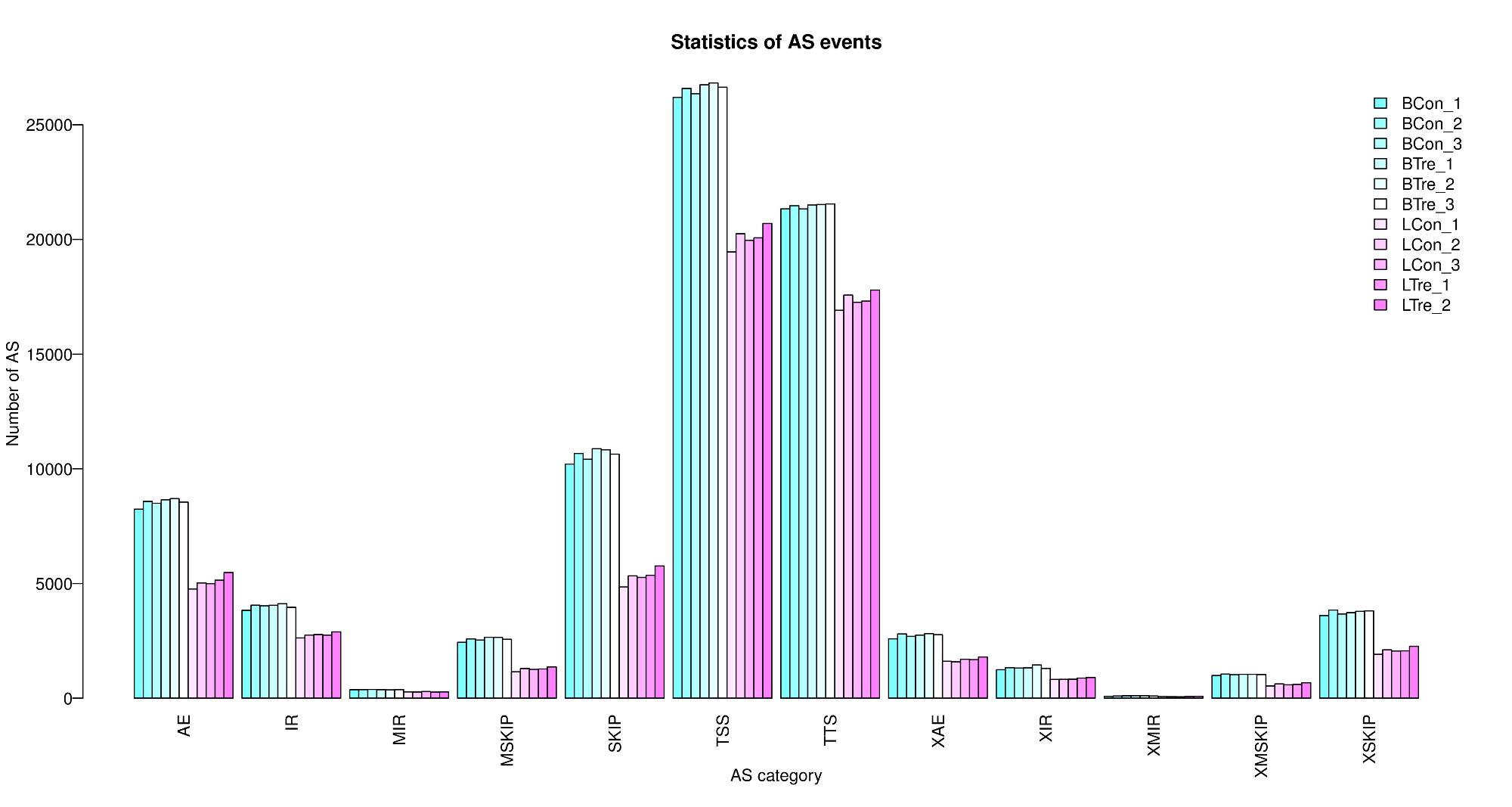
可变剪切统计图
对该物种及其相应的测序样品进行可变剪切事件的分类及统计。